近日,中國科學(xué)技術(shù)大學(xué)瞿昆教授課題組成功開發(fā)了一種基于深度學(xué)習(xí)的空間轉(zhuǎn)錄組數(shù)據(jù)分析算法,,命名為SPACEL,。該算法能夠快速高效地處理多個空間轉(zhuǎn)錄組切片數(shù)據(jù),包括準(zhǔn)確預(yù)測單個空間點(diǎn)內(nèi)的細(xì)胞類型組成,、精準(zhǔn)識別跨切片的功能性空間域,,以及有效重構(gòu)復(fù)雜組織的三維結(jié)構(gòu)。研究成果以《SPACEL: deep learning-based characterization of spatial transcriptome architectures》為題,,發(fā)表于2023年11月22日的《自然-通訊》(Nature Communications)雜志,。

空間轉(zhuǎn)錄組技術(shù)具備同時獲取基因表達(dá)水平和其空間位置信息的能力,目前被廣泛應(yīng)用于生物醫(yī)學(xué)研究的各個領(lǐng)域,。然而,,有效分析由空間轉(zhuǎn)錄組技術(shù)產(chǎn)生的大量數(shù)據(jù)仍然面臨著巨大挑戰(zhàn),例如如何準(zhǔn)確識別在基因表達(dá)和空間位置上相互關(guān)聯(lián)的細(xì)胞或點(diǎn)的集群,,即所謂的空間域,,以描繪組織切片內(nèi)的全轉(zhuǎn)錄組空間結(jié)構(gòu)。此外,,如何精確鑒定類似于腫瘤微環(huán)境這樣復(fù)雜系統(tǒng)中的功能性空間域,,以及如何整合多個切片的數(shù)據(jù)進(jìn)行上述分析也是挑戰(zhàn)之一。
瞿昆教授課題組開發(fā)的SPACEL算法包括三個核心模塊:Spoint,、Splane和Scube,,分別對應(yīng)空間轉(zhuǎn)錄組數(shù)據(jù)分析的三個關(guān)鍵任務(wù)。Spoint模塊用于預(yù)測基于測序的空間轉(zhuǎn)錄組數(shù)據(jù)(如10X Visium)中空間點(diǎn)的細(xì)胞類型組成,。Spoint采用了單細(xì)胞數(shù)據(jù)模擬的空間點(diǎn),、神經(jīng)網(wǎng)絡(luò)模型和統(tǒng)計(jì)模型的組合,,為估算真實(shí)空間轉(zhuǎn)錄組數(shù)據(jù)中的細(xì)胞類型比例提供了更穩(wěn)健和準(zhǔn)確的框架。Splane模塊則使用細(xì)胞類型組成和空間坐標(biāo)信息,,引入了對抗訓(xùn)練到圖卷積神經(jīng)模型中,,顯著減少了批次效應(yīng),從而實(shí)現(xiàn)更穩(wěn)健和高效的空間域識別,。對于包含連續(xù)切片的空間轉(zhuǎn)錄組數(shù)據(jù)集,,Scube模塊使用Splane識別的空間域坐標(biāo)進(jìn)行對齊,以構(gòu)建組織的三維結(jié)構(gòu),。Scube采用全局優(yōu)化策略來實(shí)現(xiàn)三維對齊,,同時保持整體結(jié)構(gòu)的完整性,使其能夠?qū)崿F(xiàn)更精確的對齊,。
研究人員將SPACEL應(yīng)用于11個包括10X Visium,、STARmap、MERFISH,、Stereo-seq和Spatial Transcriptomics技術(shù)的空間轉(zhuǎn)錄組數(shù)據(jù)集,,總計(jì)156個切片。SPACEL在細(xì)胞類型組成預(yù)測,、空間域識別以及組織三維結(jié)構(gòu)重構(gòu)等三個核心分析任務(wù)上表現(xiàn)出色,,明顯優(yōu)于其他同類算法。
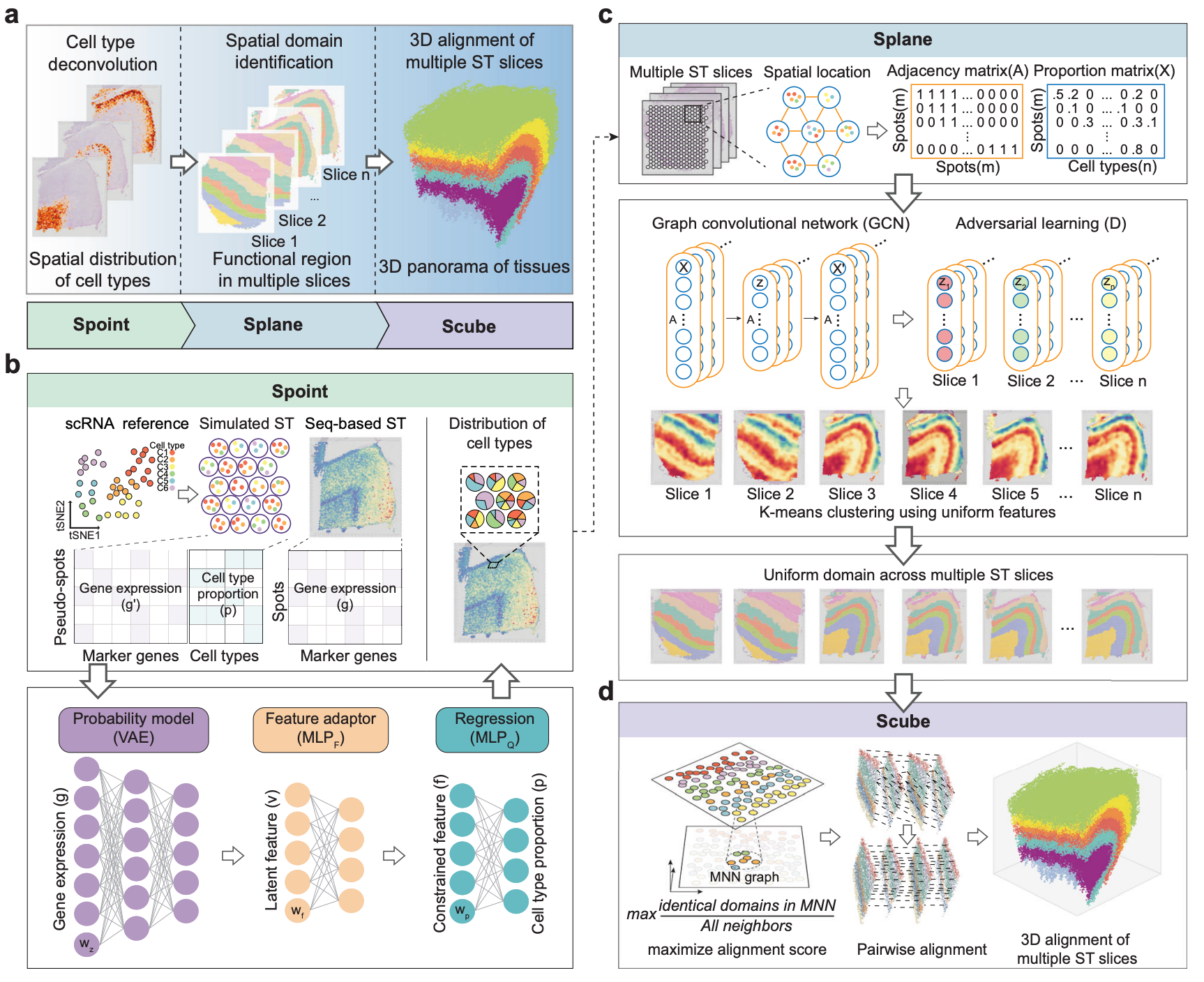
圖1.SPACEL算法的工作流程
生醫(yī)部瞿昆教授,、林俊副研究員和北京生命科學(xué)研究所黎斌研究員為本文的通訊作者,,微尺度研究中心博士生許浩和大數(shù)據(jù)學(xué)院碩士生王姝妍為本文的共同第一作者。生醫(yī)部薛天教授課題組為該工作提供了重要支持,。這一研究工作得到了基金委杰出青年基金,、國家重點(diǎn)研發(fā)計(jì)劃、基金委自然科學(xué)基金,、中國科學(xué)院基礎(chǔ)研究青年團(tuán)隊(duì)以及安徽省科技重大專項(xiàng)等多個項(xiàng)目的資助,。
論文鏈接:https://www.nature.com/articles/s41467-023-43220-3
(合肥微尺度物質(zhì)科學(xué)國家研究中心)

